Biomart MCP
Ein MCP-Server zur Schnittstelle mit Biomart
Das Model Context Protocol (MCP) ist ein offenes Protokoll, das die Bereitstellung von Kontext für LLMs durch Anwendungen standardisiert, die von Anthropic entwickelt wurden. Hier verwenden wir das MCP-Python-SDK, um einen MCP-Server zu erstellen, der über das Paket „pybiomart“ mit Biomart kommuniziert.
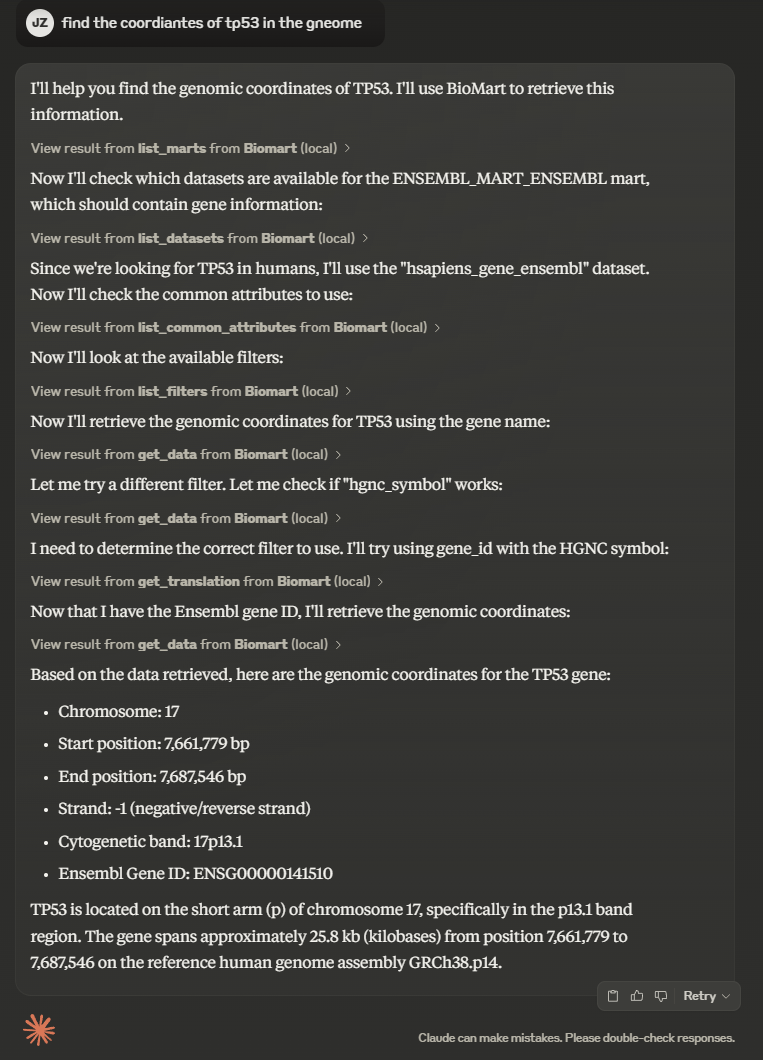
Es gibt ein kurzes Demo-Video, das den MCP-Server auf Claude Desktop in Aktion zeigt.
Installation
Installation über Smithery
So installieren Sie Biomart MCP für Claude Desktop automatisch über Smithery :
Klonen Sie das Repository
Claude Desktop
Cursor
Über den Agentenmodus von Cursor können auch andere Modelle MCP-Server nutzen, beispielsweise von OpenAI oder DeepSeek. Klicken Sie auf das Zahnrad für die Cursoreinstellungen und navigieren Sie zu MCP Fügen Sie den MCP-Server entweder zur globalen Konfiguration hinzu oder fügen Sie ihn dem Projektbereich hinzu, indem Sie .cursor/mcp.json zum Projekt hinzufügen.
Beispiel .cursor/mcp.json :
Glama
Entwicklung
Related MCP server: MCP TapData Server
Merkmale
Biomart-MCP bietet mehrere Tools zur Interaktion mit Biomart-Datenbanken:
Mart- und Dataset-Erkennung : Listen Sie verfügbare Marts und Datasets auf, um die Biomart-Datenbankstruktur zu erkunden
Attribut- und Filtererkundung : Zeigen Sie allgemeine oder alle verfügbaren Attribute und Filter für bestimmte Datensätze an
Datenabruf : Abfrage von Biomart mit bestimmten Attributen und Filtern, um biologische Daten zu erhalten
ID-Übersetzung : Konvertierung zwischen verschiedenen biologischen Kennungen (z. B. Gensymbole in Ensembl-IDs)
Beitragen
Pull Requests sind willkommen! Einige kleine Hinweise zur Entwicklung:
Wir verwenden hier absichtlich nur
@mcp.tool(), um die Kompatibilität mit Clients zu maximieren, die MCP unterstützen, wie in den Dokumenten zu sehen.Wir verwenden
@lru_cache, um Ergebnisse von Funktionen zwischenzuspeichern, die rechenintensiv sind oder externe API-Aufrufe tätigen.Wir müssen darauf achten, das Kontextfenster des Modells nicht zu sehr zu vergrößern. Beispielsweise sieht man an vielen Stellen
df.to_csv(index=False).replace("\r", ""). Diese Rückgabe im CSV-Stil ist deutlich tokeneffizienter als beispielsweisedf.to_string(), wo die meisten Token Leerzeichen sind. Beachten Sie außerdem, dass das Abrufen aller Gene aus einem Chromosom oder ähnlich umfangreiche Anfragen ebenfalls zu groß für das Kontextfenster ist.
Mögliche zukünftige Funktionen
Natürlich könnten noch viele weitere Funktionen hinzugefügt werden, einige davon gehen möglicherweise über den Namen biomart-mcp hinaus. Hier sind einige Ideen:
Fügen Sie Webscraping für Ressourcenseiten mit
bs4hinzu. Beispielsweise haben wir die Ensembl-Gen-ID für NOTCH1 erhalten. In einigen Fällen wäre es möglicherweise nützlich, die gesammeltenComments and Description Text from UniProtKBvon der Seite auf UCSC abzurufen.$...$