バイオマートMCP
BiomartとインターフェースするMCPサーバー
モデルコンテキストプロトコル(MCP)は、 Anthropicが開発したLLMにアプリケーションがコンテキストを提供する方法を標準化するオープンプロトコルです。ここでは、MCP Python SDKを使用して、 pybiomartパッケージを介してBiomartとインターフェースするMCPサーバーを作成します。
Claude Desktop 上で動作している MCP サーバーを示す短いデモ ビデオがあります。
インストール
Smithery経由でインストール
Smithery経由で Biomart MCP for Claude Desktop を自動的にインストールするには:
リポジトリをクローンする
クロードデスクトップ
カーソル
Cusrorのエージェントモードを使用すると、OpenAIやDeepSeekなどのMCPサーバーを他のモデルでも利用できます。カーソル設定の歯車をクリックしてMCPに移動し、MCPサーバーをグローバル設定に追加するか、 .cursor/mcp.jsonプロジェクトに追加してプロジェクトスコープに追加してください。
例.cursor/mcp.json :
グラマ
発達
Related MCP server: MCP TapData Server
特徴
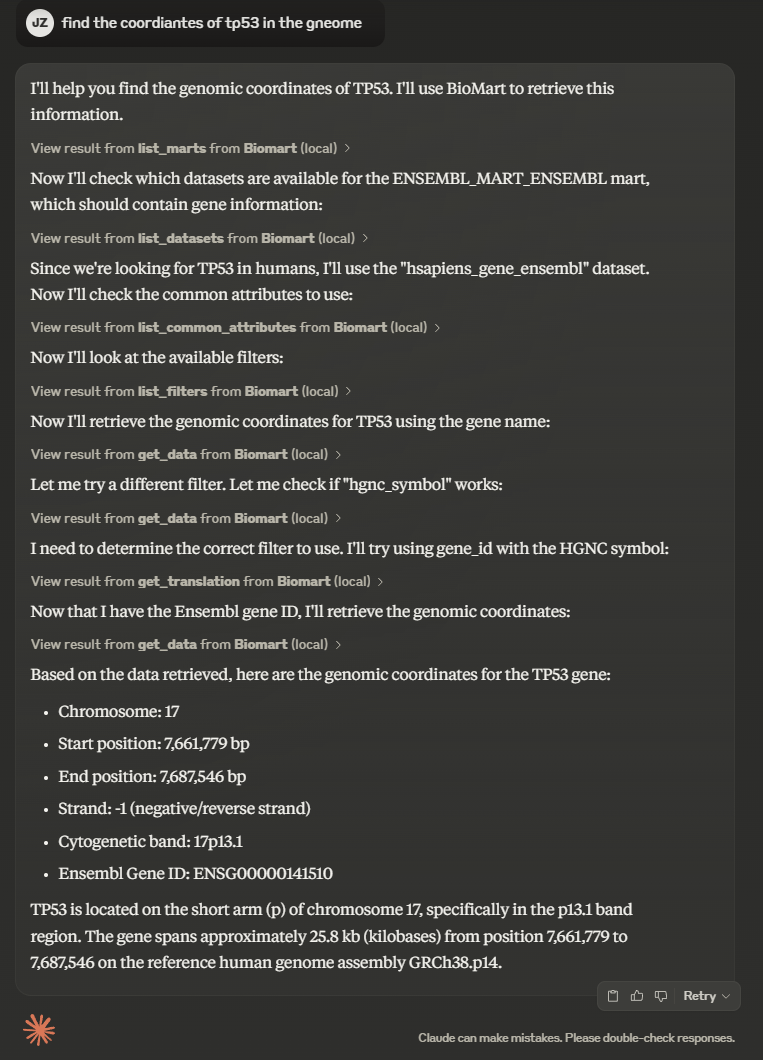
Biomart-MCP は、Biomart データベースと対話するためのいくつかのツールを提供します。
Mart とデータセットの検出: Biomart データベース構造を探索するために利用可能な mart とデータセットを一覧表示します
属性とフィルターの探索: 特定のデータセットの共通または利用可能なすべての属性とフィルターを表示します。
データ取得: 特定の属性とフィルターを使用して Biomart をクエリし、生物学的データを取得します。
ID 変換: 異なる生物学的識別子 (例: 遺伝子シンボルから Ensembl ID) 間の変換
貢献
プルリクエストは大歓迎です!開発に関するちょっとしたメモ:
ここでは意図的に
@mcp.tool()のみを使用しています。これは、ドキュメントに記載されているように、MCP をサポートするクライアントとの互換性を最大化するためです。計算コストが高い関数や外部 API 呼び出しを行う関数の結果をキャッシュするために、
@lru_cacheを使用しています。モデルのコンテキストウィンドウを拡張しないように注意する必要があります。例えば、多くの場所で
df.to_csv(index=False).replace("\r", "")見られます。このcsv形式の戻り値は、トークンの大部分が空白であるdf.to_string()のような関数よりもトークン効率がはるかに高くなります。また、染色体からすべての遺伝子を取得するなど、大規模なリクエストはコンテキストウィンドウに対して大きすぎることにも注意してください。
将来の潜在的な機能
もちろん、他にも追加できる機能はたくさんあり、中にはbiomart-mcpという名前の範囲を超えるものもあるかもしれません。いくつかアイデアをご紹介します。
bs4を使ってリソース サイトの Web スクレイピングを追加します。たとえば、NOTCH1 の Ensembl 遺伝子 ID を取得した場合、場合によっては、 UCSC のページからComments and Description Text from UniProtKBを取得することが役に立つ可能性があります。$...$